Le système CRISPR/Cas9 : Pour la compréhension du vivant et l’innovation variétale
Cécile Collonnier

Un ciseau moléculaire d’origine bactérienne
L’étude du système CRISPR/Cas9 a impliqué de nombreuses équipes dans le monde, parmi lesquelles les chercheurs français Philippe Horvath et Isabelle Charpentier ont joué un rôle déterminant (Lander, 2016).
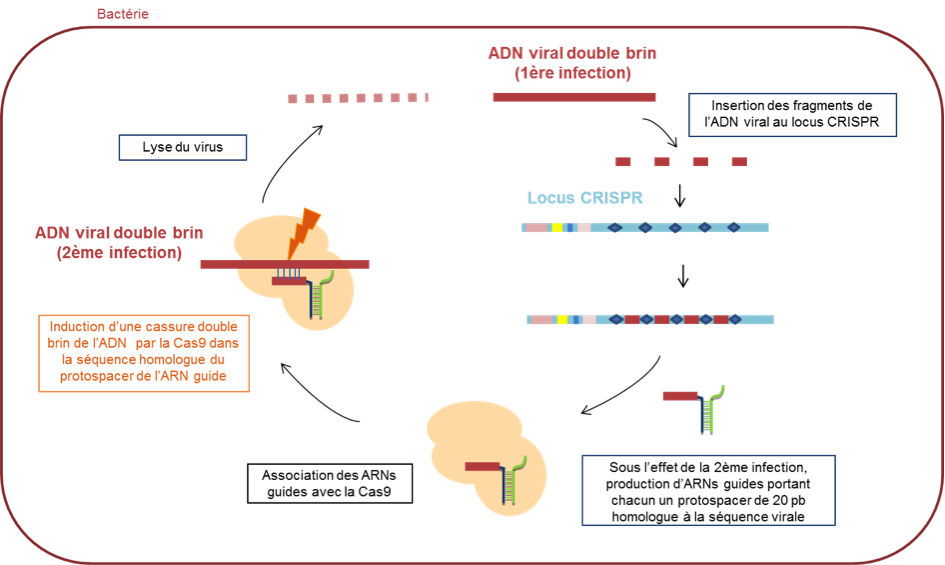
Le système CRISPR/Cas9[1] est à l’origine un mécanisme de défense immunitaire développé par certaines bactéries pour lutter contre l’invasion d’ADN exogène, principalement des virus. Lors de leur première invasion dans la cellule, les séquences de ces virus sont détectées et intégrées en fragments de 20 nucléotides dans une zone du génome bactérien appelée locus CRISPR (Figure 1). Ce locus est une sorte de mémoire des séquences virales ayant infecté la bactérie. Si l’un de ces virus se présente à nouveau, le locus CRISPR est activé et produit de petits ARNs non codants, appelés ARNs guides, contenant chacun un des fragments de 20 pb de la séquence du virus (le protospacer). Chaque ARN guide s’associe à une protéine capable de couper l’ADN, la nucléase Cas9, qu’il guide ensuite jusqu’à la région du virus dont il porte lui-même une copie. L’action des protéines Cas9 qui coupent chaque séquence aboutit à la lyse du virus complet (Horvath and Barrangou, 2010).
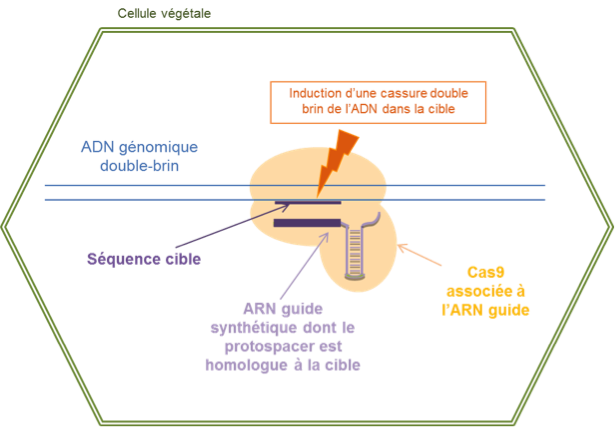
Ce système naturel a été adapté pour induire de manière ciblée des cassures dans le génome des organismes eucaryotes[2]. Les ARNs guides bactériens sont remplacés par des ARNs guides synthétiques où la séquence du protospacer est modifiable de manière à induire une coupure de l’ADN dans n’importe quelle séquence cible (Figure 2) (Jinek et al., 2012). Chez les plantes, les gènes codant l’ARN guide et la Cas9 sont simultanément introduits dans la cellule où ces deux éléments s’associent pour aller couper la séquence ciblée.
Pourquoi induire des coupures dans le génome ?
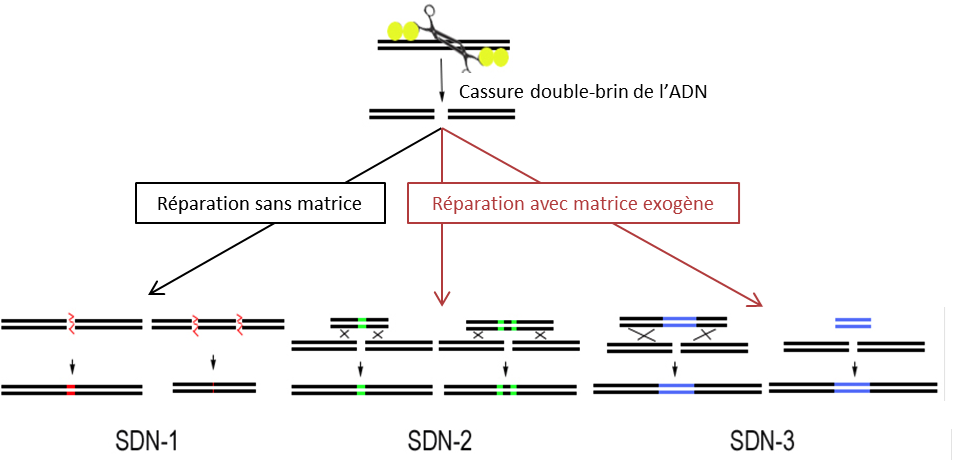
Lorsqu’une cassure de l’ADN est générée, elle est immédiatement réparée par la machinerie cellulaire. De temps en temps, une erreur se produit ce qui permet de créer une mutation dans la séquence cible (Figure 3 : type SDN1). Si au moment de la coupure, on fournit à la cellule une matrice d’ADN comportant une séquence d’intérêt, celle-ci peut également être utilisée pour réparer la cassure, ce qui aboutit à son intégration dans la cible. Cette séquence d’intérêt peut être soit une version allélique d’un gène qui vient remplacer l’allèle existant (Figure 3 : type SDN2), soit un transgène apportant un nouveau caractère (Figure 3 : type SDN3). On peut donc ainsi modifier de manière contrôlée presque n’importe quelle zone du génome.
De grands enjeux pour la recherche

Le système CRISPR/Cas9 est un outil facile à mettre en œuvre, adaptable, rapide, peu onéreux et d’une grande efficacité. Il permet de créer des mutants à la demande pour étudier les fonctions des gènes, leurs interactions ou leur régulation dans n’importe quelle voie métabolique (Piatek et al., 2015). Toutes ces utilisations potentielles ouvrent un champ immense de progrès dans la compréhension des mécanismes qui régissent le vivant.
Un énorme potentiel pour la création variétale
L’intégration du système CRISPR/Cas9 dans les schémas de sélection devrait donner aux obtenteurs la possibilité de mieux contrôler et d’accélérer la création de nouvelles variétés, et ouvre des perspectives presqu’illimitées de combinaisons de nouveaux allèles d’intérêt identifiés dans des espèces même sexuellement incompatibles. L’utilisation de ce système pourrait également rendre plus accessibles des objectifs de sélection particuliers, comme : la modification du génome d’espèces polyploïdes, la sélection d’allèles récessifs, l’élimination d’allèles défavorables…

De plus, le système CRISPR/Cas9, qui s’affranchit des barrières sexuées et du fardeau génétique lié aux hybridations, pourrait permettre de faciliter l’exploitation des ressources génétiques. En effet, les collections sont une source d’informations très importante pour l’identification de gènes d’intérêt qui pourraient, soit être insérés directement dans le génome des variétés, soit servir à l’identification de gènes homologues potentiellement modifiables chez les espèces cultivées.
De nombreuses applications agronomiques
Le système CRISPR/Cas9 a déjà été appliqué à une large gamme d’espèces végétales (Sovová et al., 2016). On compte ainsi, des tolérances aux maladies (à l’oïdium chez le blé (Wang et al., 2014), au feu bactérien chez le riz (Zhou et al., 2016) et à des virus chez le concombre (Chandrasekaran et al., 2016)), et des modifications de profils de maturation chez la tomate (Ito et al., 2015). Dans le futur, de nombreuses autres applications pourraient voir le jour, tels des tolérances aux stress abiotiques (sécheresse, salinité, température,…), des facteurs de qualité améliorés (composés nutritionnels), des caractéristiques physiologiques modifiées (biomasse, floraison accélérée, stérilité mâle inductible), la production de molécules d’intérêt, …
Remerciements pour les crédits photographiques à François Maclot, bio ingénieur et botaniste de la Faculté d’Agronomie de Gembloux (Gx-ABT), Université de Liège.
A lire …
– Chandrasekaran, J., Brumin, M., Wolf, D., Klap, C., Sherman, A. and Arazi, T. Development of broad virus resistance in non-transgenic cucumber using CRISPR / Cas9 technology (2016) Molec. Plant Pathol. 17 (7), 1140-1153.
– Horvath, P. and Barrangou, R. (2010) CRISPR/Cas, the Immune System of Bacteria and Archaea. Science 327, 167-170.
Ito, Y., Nishizawa-yokoi, A., Endo, M. and Mikami, M. (2015) Biochemical and Biophysical Research Communications CRISPR / Cas9-mediated mutagenesis of the RIN locus that regulates tomato fruit ripening. Biochem. Biophys. Res. Commun., 1–7.
– Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J.A. and Charpentier, E. (2012) A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science (80-. ). 337, 816–821.
Lander, E.S. (2016) The Heroes of CRISPR. Cell 164, 18–28.
– Piatek, A., Ali, Z., Baazim, H., Li, L., Abulfaraj, A., Al-Shareef, S., Aouida, M. and Mahfouz, M.M. (2015) RNA-guided transcriptional regulation in planta via synthetic dCas9-based transcription factors. Plant Biotechnol. J. 13, 578–589.
– Podevin, N., Davies, H. V., Hartung, F., Nogué, F. and Casacuberta, J.M. (2013) Site-directed nucleases: A paradigm shift in predictable, knowledge-based plant breeding. Trends Biotechnol. 31, 375–383.
– Sovová, T., Kerins, G., Demnerova, K. and Ovesna, J. Genome Editing with Engineered Nucleases in Economically Important Animals and Plants : State of the Art in the Research Pipeline. Curr. Issues Mol. Biol. 21, 41–62.
– Wang, Y., Cheng, X., Shan, Q., Zhang, Y., Liu, J., Gao, C. and Qiu, J. (2014) Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat. Biotechnol., 1–6.
– Zhou, D., Chen, W., Lin, Z., et al. (2016) Pedigree-based analysis of derivation of genome segments of an elite rice reveals key regions during its breeding. Plant Biotechnol. J. 14, 638–648.
[1] CRISPR/Cas9 : Clustered Regularly Interspaced Short Palindromic Repeats / CRISPR-associated protein)
[2] Organismes uni- ou pluricellulaires dont les cellules possèdent un noyau renfermant tout ou partie de leur l’ADN. Ils comprennent les protozoaires, les plantes et champignons, ainsi que les animaux.
